Indice del volumen Volume index
Comité Editorial Editorial Board
Comité Científico Scientific Committee
- Ácido 2,5-dihidroxibenzoico (DHB) para analizar péptidos, proteínas, lípidos y oligosacáridos.
- Ácido 3,5-dimetoxi-4-hidroxicinámico (Ácido sinapínico) para analizar péptidos, proteínas y glicoproteínas.
- Ácido
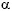 -cyano-4-hidroxicinámico (HCCA) para analizar péptidos, proteínas, lípidos y oligonucleótidos.
-cyano-4-hidroxicinámico (HCCA) para analizar péptidos, proteínas, lípidos y oligonucleótidos.
- Importación de archivos ejecutados con el espectrómetro de masas de Bruker.
- Visualización de los espectros.
- Identificación de espectros desconocidos por comparación con espectros de referencia de la base de datos para poder realizar la identificación de los microorganismos.
- Manejo de la información de la muestra: origen, condiciones de crecimiento y datos demográficos.
- Aplicación de métodos de clustering y realización de dendogramas para el agrupamiento de microorganismos.
- Creación de bases de datos propias para así identificar microorganismos con unas características concretas.
IMPACTO DE LA METODOLOGÍA MALDI-TOF EN LA IDENTIFICACIÓN CLÍNICA DE AGENTES INFECCIOSOS.
Gabriel A. March, José M. Eiros
Hospital Clínico Universitario y Facultad de Medicina de Valladolid
Valladolid. España
gmr810 @ hotmail.com
Rev Electron Biomed / Electron J Biomed 2012;1:60-65
Comentario de la revisora Maria Ángeles Mantecón PhD. Sección de Microbiología Clínica. Complejo Asistencial Universitario de Burgos. España.
Comentario de la revisora Silvia Albillos PhD. Jefe de Área de Investigación del Instituto de Biotecnología de León (INBIOTEC). León. España.
RESUMEN:
La denominación "MALDI" deriva de las siglas de las palabras en inglés Matrix-Assisted Laser Desorption/Ionization (desorción/ionización láser asistida por matriz) y "TOF" que alude al detector de iones que se acopla al MALDI, que es del tipo de tiempo de vuelo (Time of Flight).
Se trata de una metodología de ionización suave utilizada en espectrometría de masas que representa una técnica analítica instrumental de alta sensibilidad capaz de identificar cuali y cuantitativamente cualquier tipo de mezclas de sustancias.
Es una técnica muy utilizada en proteómica para la identificación de microorganismos mediante el método de la huella peptídica. De manera esquemática describiremos en la presente revisión los aspectos relativos a su aplicación en Microbiología Clínica
PALABRAS CLAVE: MALDI, Microbiología Clínica
SUMMARY:
The term "MALDI" is the abbreviation of Matrix-Assisted Laser Desorption / Ionization and "TOF" which refers to the ion detector coupled to MALDI, which is based on the time of flight of the ions.
MALDI is a soft ionization method used in mass spectrometry analytical technique that represents a highly sensitive instrument capable of qualitatively and quantitatively identifying any mixtures of substances.
It is a technique widely used in proteomics for the identification of microorganisms by the method of the peptide fingerprint. Schematically we describe in this review the most relevant aspects of its application in Clinical Microbiology.
KEYWORDS: MALDI, Clinical Microbiology
INTRODUCCIÓN
MALDI-TOF es una metodología de ionización suave utilizada en espectrometría de masas que representa una técnica analítica instrumental de alta sensibilidad capaz de identificar cuali- y cuantitativamente cualquier tipo de mezclas de sustancias. Asimismo, esta técnica también permite determinar la masa molecular de un compuesto, y la de los diversos fragmentos que resultan de la rotura del mismo, proporcionando una información muy valiosa sobre la estructura de la molécula.
La denominación "MALDI" deriva de las siglas de las palabras en inglés Matrix-Assisted Laser Desorption/Ionization (desorción/ionización láser asistida por matriz) y "TOF" alude al detector de iones que se acopla al MALDI, que es del tipo de tiempo de vuelo (Time of Flight).
Su desarrollo se remonta a finales de la década de los 80, cuando se observó que utilizando una fuente de láser ultravioleta y embebiendo la muestra en una matriz formada por moléculas aromáticas se conseguía una ionización blanda, proporcionando la fragmentación adecuada para la identificación de moléculas lábiles como proteínas, péptidos, azúcares y oligonucleótidos1,2.
El MALDI-TOF es una técnica muy utilizada en proteómica para la identificación de microorganismos mediante el método de la huella peptídica3-6. En este método, las proteínas desconocidas en estudio son hidrolizadas en pequeños péptidos cuyas masas absolutas se determinan mediante un espectrómetro de masas. La huella de tamaños de péptidos obtenida, que es única para cada microorganismo, es comparada con las huellas de microorganismos conocidos presentes en la base de datos de tal forma que la huella problema se puede asociar estadísticamente con la huella más semejante y realizar así la identificación del microorganismo7,8.
Las proteínas que conforman la huella peptídica de cada microorganismo son las ribosomales debido a que se comportan como proteínas estructurales y a que se mantienen una estructura altamente conservada a lo largo del tiempo. De manera complementaria también se incluyen proteínas de membrana y de origen intracelular9,10.
De manera esquemática describiremos los siguientes apartados relativos a su aplicación en Microbiología Clínica.
INSTRUMENTACIÓN11
1. ESPECTROMETRO DE MASAS
La espectrometría de masas, como se ha avanzado, es una técnica analítica que nos permite identificar y cuantificar la masa molecular de iones libres, lo que requiere trabajar en condiciones de alto vacio. Los espectrómetros de masas están formados por tres elementos básicos. En primer lugar la "fuente de iones", donde a partir de la muestra se forma un haz de iones en estado gaseoso. En segundo término el "separador de masas" o "tubo de vuelo", que separa los iones formados en función de su relación "masa/carga" (m/z) y en tercera instancia el "detector" de los iones previamente separados.
La información final que nos proporciona esta técnica es la relación m/z de los distintos iones formados y en qué cantidad relativa están presentes.
2. PRINCIPIO DE IONIZACIÓN-MALDI
Para conseguir una ionización, la muestra previamente depositada sobre la placa metálica que contiene los biopolímeros que pretendemos identificar, se pone en disolución con una gran cantidad de una sustancia matriz que debe contener en su estructura anillos aromáticos. Después se evapora el disolvente a temperatura ambiente produciéndose la co-cristalización de la muestra y matriz de tal forma que, las moléculas de la muestra quedan incorporadas dentro de la estructura de los cristales de la matriz. Esta etapa del procedimiento resulta esencial para conseguir una posterior desorción/ionización eficiente.
A continuación, la placa es introducida en la cámara de alto vacio, donde la superficie cristalina de la muestra es expuesta a disparos de radiación láser de longitud de onda del UV, con lo que las moléculas orgánicas aromáticas de la matriz absorben una gran cantidad de energía por excitación de los electrones  (electrones de dobles y triples enlaces) produciéndose la sublimación del analito y matriz depositado sobre la placa.
(electrones de dobles y triples enlaces) produciéndose la sublimación del analito y matriz depositado sobre la placa.
Ya en fase gaseosa, la estabilización de estas moléculas aromáticas tiene lugar por adición de protones que, en parte son captados por los analitos de la muestra, que en nuestro caso son mayoritariamente las proteínas ribosomales de las bacterias, generándose fragmentos de proteínas con carga positiva.
Seguidamente, mediante un electrodo situado a unos pocos mm en frente de la placa se impone un campo eléctrico que acelera los iones formados desde las proximidades de la muestra hacia el analizador de masas, donde como sabemos los iones formados se separarán en función de su relación masa/carga (m/z). Para que se produzca una ionización eficiente de la muestra los pulsos del láser deben ser de energía entre 106 y 107 W/cm cuadrados, y de una duración de 1 a 5 ns. Cada muestra se expone a 240 disparos.
3. ANÁLISIS DE LAS MASAS DE LOS IONES CON EL ANALIZADOR DE TIEMPO DE VUELO
El analizador de tiempo de vuelo es básicamente un tubo (de 1 a 4 metros) donde entran los iones generados por el láser siguiendo una trayectoria lineal. En estas condiciones todos los iones que entran en el tubo tienen la misma energía cinética, que podemos escribir como:
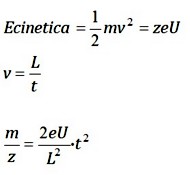
U=Campo Eléctrico (constante)
e=carga del electrón (constante)
Z=carga del ión
L=Longitud del tubo (constante)
Donde la relación m/z es proporcional al cuadrado del tiempo de vuelo, por tanto al ser menor la relación m/z de los iones, menor es el tiempo necesario para recorrer el tubo de longitud L, es decir, los iones de menor m/z "vuelan" más rápido. En la práctica estos tiempos varían desde unos pocos microsegundos a 100 microsegundos.
4. PODER DE RESOLUCIÓN
Una importante cualidad del analizador de masas es el poder de resolución. Representa la capacidad del espectrómetro para distinguir entre iones de distinta masa/carga próxima. Si m1 es la m/z del pico que nos interesa cuantificar, y m2 la del pico más próximo que podemos diferenciar de m1, matemáticamente la resolución se define como:
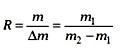
De este modo, un espectrómetro con un poder de resolución de 20000 (que es el que tiene poseen los MALDIs incorporados a la red asistencial de diagnóstico) nos permite diferenciar entre dos picos de masas de 200000 y 200010 Da, pero no entre picos más próximos.
En los analizadores de tiempo de vuelo la resolución se calcula con la misma fórmula pero a partir de un solo pico considerando que el incremento de m de los dos picos (m2-m1) es la anchura del pico a la mitad de la altura.
5. DETECTOR DE IONES
El detector de iones consiste básicamente en una superficie de materiales semiconductores sobre la que impactan los iones positivos (procedentes de los analitos) que salen separados del tubo analizador. Como consecuencia de cada impacto contra la superficie semiconductora se emiten electrones, que por la geometría del detector, se proyectan contra otros puntos de la superficie del detector provocando la emisión de un mayor número de electrones secundarios (los electrones primarios son los que resultan del impacto de los iones positivos, mientras que los electrones secundarios son los que se generan por el impacto de los electrones primarios). Por este motivo estos detectores se conocen con el nombre de "Sistemas Multiplicadores de Electrones Secundarios".
De esta forma se genera un "output de electrones" o pulso de electrones, cuya carga (es decir número de electrones) depende del número de iones con la misma masa que impactan en un mismo instante contra la superficie detectora.
Como los iones salen del tubo analizador separados en el tiempo por orden creciente de su m/z para cada disparo láser tendremos una serie de output de carga en función del tiempo que tarde en llegar al detector que, aplicando la fórmula 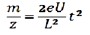 el tiempo se transforma en la relación m/z de los distintos iones formados.
el tiempo se transforma en la relación m/z de los distintos iones formados.
La carga de cada "output", que es proporcional al número de iones que en un mismo instante impactan contra el detector, se designa como "intensidad". La representación de la intensidad frente a la relación m/z es lo que se conoce como espectro de masas. Si z=1, como es habitual en la ionización blanda de la técnica MALDI, en el espectro de masas se representa la intensidad frente a la masa.
En el espectro de masas, el pico de mayor intensidad recibe el nombre de "pico base", y el de mayor masa se llama "pico molecular", ya que suele representar la molécula sin fragmentar. Normalmente las intensidades se expresan en términos relativos como porcentaje del pico base, por ello la escala de intensidades va de cero a 100. Dependiendo de como esté calibrado el equipo, los valores de las masas de los iones formados en la técnica Maldi 2000 a 20000 Da.
Debido a que con un disparo obtenemos un espectro de masas, podemos asumir que con los 240 disparos realizados sobre la misma muestra se obtienen 240 espectros. Para conseguir un único espectro, se representan todos los picos obtenidos en un solo espectro de masas sumando las intensidades de los picos idénticos.
6. ROTURA DE IONES INESTABLES 12
Un aspecto relevante es el que alude a la descomposición de iones inestables. En este ámbito las siglas PSD derivan del efecto "Post Source Decay", que consiste en la descomposición de iones antes de que éstos lleguen al detector. A estos iones inestables se les denomina iones metaestables y su análisis proporciona una información útil sobre la estructura del ión original. La aparición de iones metaestables es un proceso inevitable que se debe a la elevada excitación de la matriz por parte del láser y al voltaje de aceleración aplicado. Se estima que del 10 al 50% de los iones formados se descomponen.
Como regla general, en la descomposición de un ión metaestable se forman dos fragmentos, un fragmento cargado positivamente y otro fragmento neutro, sin carga.
El tiempo en el cual tiene lugar la descomposición determina la calidad de la ionización de la muestra. Una temprana descomposición, anterior a la entrada de los iones en el tubo, produce unos iones que se separan en función de su m/z, y en consecuencia obtenemos señales que nos proporcionan información útil para deducir la masa del ión inicial.
Por el contrario, si la descomposición tiene lugar dentro del tubo de vacío, los fragmentos formados adquieren diferentes energías cinéticas y la secuencia con la que salen estos fragmentos del tubo no coincide con la secuencia de su m/z inicial. De esta forma se obtienen señales que no aportan información y además se deteriora la relación señal-ruido del espectro.
Debido a esta descomposición de los iones metaestables, se forman especies neutras que debemos evitar que impacten contra el detector, ya que generarían picos intercalados entre los picos de los iones.
Para ello, a la salida de los tubos se ha incorporado un reflector que mediante los potenciales eléctricos adecuados, tiene la función de cambiar la dirección de los iones positivos con lo que el detector está ubicado en una posición distinta de la salida del tubo y recibe el nombre de detector reflector. Las masas neutras no se ven afectadas por los potenciales del reflector por lo que siguen con su trayectoria lineal, y si interesa pueden ser detectadas por otra superficie detectora denominada detector lineal, ubicado a la salida del tubo.
7. VARIABLES ANALÍTICAS
Entre las variables analíticas instrumentales cabe aludir a la energía y número de disparos del láser, los voltajes del detector y de aceleración así como al tiempo de extracción de los iones.
Por lo que hace referencia a las variables analíticas experimentales cabe comentar diferentes aspectos de acuerdo con Giebel et al13
La concentración óptima bacteriana para una correcta identificación por MALDI-TOF es de 0,13mg/mL de matriz. Cuando la concentración es demasiado baja el instrumento es incapaz de detectar picos y si es muy elevada se producen demasiados picos dominantes, imposibilitando la identificación en ambos casos, por lo que la cantidad de muestra que se deposita en la placa metálica es crucial para la identificación por MALDI-TOF. Estos aspectos han sido también objetivados por Liu et al13.
No hay diferencias sustanciales entre espectros de masas obtenidos a partir del mismo microorganismo cultivado en diferentes medios de cultivo. Cabe apuntar como excepción que en el caso de hongos filamentosos, los espectros obtenidos de hongos que han crecido en medios que promueven la formación de conidias (agar malta, agar papa dextrosa), ofertan un espectro mucho mejor que el de los hongos que han crecido en medios convencionales.
Con respecto al tiempo de crecimiento bacteriano, en general el número de picos obtenidos se va incrementando durante las primeras 28 horas de crecimiento del microorganismo. La intensidad de los picos varía según el tiempo de crecimiento de los microorganismos.
Existen diferentes matrices con diferentes aplicaciones analíticas. Entre ellas destacan:
La matriz HCCA es usada para la detección de picos de baja masa pudiendo llegar a la detección de iones de 10 kDa. La matriz DHB también es útil para la detección de picos de baja masa. La matriz ácido sinapínico es usada para la detección de picos de elevada masa.
De todas las matrices, la HCCA es la más efectiva para proporcionar picos de alta calidad y espectros reproducibles. Es la que nosotros aplicamos en el laboratorio de Microbiología Clínica.
Con respecto a la preparación de la muestra, las muestras tratadas con lisozima presentan unos espectros cuyos picos más intensos tienen una mejor relación señal-ruido. El uso de surfactantes aumenta el rango de masas del espectro obtenido y aumenta la solubilidad de proteínas hidrofóbicas15.
Con el protocolo de extracción que adoptamos en nuestro laboratorio se consigue aumentar la fiabilidad y reproducibilidad de los espectros realizados. El metanol se utiliza para precipitar las proteínas. La función del ácido fórmico y acetonitrilo es evitar que se formen cristales a un pH >4.
8. UTILIDADES DEL PROGRAMA MALDI BIPOTYPER 2.011
A continuación se detallan las diferentes utilidades del programa MALDYBIOTYPER 2.0.
REFERENCIAS
1.- Tanaka K, Ido Y, Akita S, Yoshida Y, Yoshida T. Detection of high mass molecules by laser desorption time-of-flight mass spectrometry. Proceed 2nd Japan-China Joint Symp Mass Spectrom 1987; 185-188.
2.- Tanaka K, Waki H, Ido Y, Akita, S, Yoshida Y, Yoshida T. Protein and polymer analyses up to m/z 100 000 by Laser Ioniation Time-of-Flight Mass spectrometry. Rapid Comunn Mass Spectrom 1988; 2:151-153.
3.- Sauer S, Freiwald A, Maier T, Kube M, Reinhardt R, Kostrzewa M et al. Classification and identification of bacteria by mass spectrometry and computational analysis. PLoS One 2008; 3:e2843:1-10
4.- Freiwald A, Sauer S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nat Protoc 2009; 4:732-742
5.- Sauer S, Kliem M. Mass spectrometry tools for the classification an identification of bacteria. Nat Rev Microbiol 2010; 8:74-82
6.- Ho YP, Reddy PM. Identification of pathogens by mass spectrometry. Clin Chem. 2010; 56:525-536
7.- Demirev PA, Ho YP, Ryzhov V, Fenselau C. Microorganism identification by mass spectrometry and protein database searchers. Anal Chem 1999; 71:2732-2738
8.- Keys CJ, Dare DJ, Sutton H, Wells G, Lunt M, McKenna T et al. Compilation of a MALDI-TOF mass spectral database for the rapid screening and characterisation of bacteria implicated in human infectious diseases. Infect genet Evol 2004; 4:221-242
9.- Conway GC, Smole SC, Sarracino DA, Arbeit RD, Leopold PE. Phyloproteomics: Species Identification of Enterobacteriaceae Using Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Mass Spectrometry. J Mol Microbiol biotechnol 2001; 3:103-112
10.- Dalluge JJ. Matrix-assisted laser desorption ionization-mass spectrometry (MALDI-MS). Anal Bioanal Chem 2002; 372:18-19
11.- MALDI Theory Mass Spectrometry Bruker Daltonics®
12.- Spengler B, Kirsch D, Kaufmann, R. Metastable decay of peptides and proteins in matrix-assisted laser-desorption mass spectrometry. Rapid Commun Mass Spectrom 1991; 5:198-202
13.- Giebel R, Worden C, Rust SM, Kleinheinz GT, Robbins M, Sandrin TR. Microbial fingerprinting using matrix-assisted laser desorption ionization time-of-flight mass spectrometry (MALDI-TOF MS) applications and challenges. Adv Appl Microbiol 2010;71:149-184
14.- Liu H, Du Z, Wang J, Yang R. Universal sample preparation method for characterization of bacteria by matrix-assisted laser desorption ionization-time of flight mass spectrometry. Appl. Environ. Microbiol. 2007; 73:1899-1907.
15.- Meetani MA, Voorhees KJ. MALDI Mass Spectrometry Analysis of High Molecular Weight Proteins from Whole bacterial Cells: Pretreatment of Samples with Surfactants. J Am Soc Mass Spectrom 2005; 16:1422-1426
CORRESPONDENCIA:
Dr. Gabriel Alberto March Rosselló
Plaza Trinidad número 5.
47003 Valladolid
gmr810 @ hotmail.com
