Indice del volumen Volume index
Comité Editorial Editorial Board
Comité Científico Scientific Committee
MICRO RNA EXTRACELULARES EN EL CÁNCER. POTENCIALES BIOMARCADORES DE DIAGNÓSTICO Y DESARROLLO
Raquel Alcaraz1, Patricia Saiz-López2, Pilar Muñiz3,
Carlos Garcia-Girón4.
1Unidad de Investigación, 2Anatomía Patológica y 4Oncología Médica del Hospital Universitario de Burgos;
3Departamento de Biotecnología y Ciencia de los Alimentos de la Universidad de Burgos
Burgos. España
Email: ralcaraz @ hubu.es
Rev Electron Biomed / Electron J Biomed 2017;1:51-58.
Comentario del revisor Daniel Alameda Serrano, PhD. Programa de Tumores Sólidos. Centro de Investigación Médica Aplicada (CIMA), Pamplona. España.
Comentario de la revisora Prof. Victoria Valls i Belles, PhD. Unidad Predepartamental de Medicina. Facultad de Medicina, Universidad de Castellón. España
RESUMEN
Los microRNA (miRNA) son pequeñas moléculas de ácido ribonucleico, con funciones reguladoras de la expresión genética implicadas en la diferenciación, proliferación, apoptosis y metabolismo celular. Su expresión aberrante está asociada a diferentes patologías entre ellas la oncogénesis.
Los miRNAs pueden detectarse en suero, plasma, orina y saliva de forma estable y reproducible, por lo que pueden ser útiles como biopsia clínica para la diagnosis del cáncer. En esta revisión, se resumen los estudios enfocados a la identificación de miRNAs asociados a diferentes tipos de cáncer.
PALABRAS CLAVE: microRNA, biopsia líquida, cáncer
ABSTRACT
MicroRNAs (miRNA) are small molecules of ribonucleic acid, with regulatory functions of gene expression involved in differentiation, proliferation, apoptosis and cellular metabolism. Its aberrant expression is associated with different pathologies including oncogenesis.
The miRNAs can be detected in serum, plasma, urine and saliva which is stable and reproducible, they can be useful as a clinical biopsy for the diagnosis of cancer. In this review, we summarize studies focused on the identification of miRNAs associated with different types of cancer.
KEY WORDS: microRNA, liquid biopsy, cancer
INTRODUCCIÓN
Entre los diferentes tipos de cáncer, los de piel, colon, pulmón y hematopoyéticos se encuentran entre los diez más comunes en la sociedad occidental1, y, aun siendo accesibles quirúrgicamente los tumores sólidos, presentan un mal pronóstico debido a una detección tardía. Estos cuatro tipos de tumores parten de tejidos con variabilidad en cuanto a su origen y composición celular (ectodérmico mesodérmico y endodérmico), sin embargo el interés en dar una respuesta traslacional al conjunto de las enfermedades cancerosas hace que la evolución del proceso de todas ellas sea muy similar.
En la actualidad una de las áreas prioritarias en el área oncológica es encontrar nuevos métodos para una detección temprana del cáncer, como principal aproximación para su prevención, a través de técnicas no invasivas, donde destaca la búsqueda de biomarcadores en biopsias líquidas. En este sentido, el desarrollo de biomarcadores obtenidos a partir de plasma y saliva cuya cuantificación permita detectar a tiempo el desarrollo y grado de evolución del tumor comienza a ser una realidad. Por ello el estudio de microRNAs (miRNA) presentes en fluidos biológicos, pueden ser utilizados como biomarcadores pronóstico del cáncer.
Estructuralmente los miRNA, son transcritos de RNA no codificante (ncRNA) de pequeño tamaño (19-22 nucleótidos), que desempeñan un papel importante en la regulación de la actividad celular a nivel transcripcional2. Los miRNA están implicados en procesos de diferenciación, proliferación y supervivencia celular3-4. Actúan como pequeñas guías moleculares en el silenciamiento del RNA mediante regulación negativa de la expresión de genes, mediante procesos de degradación de sus mRNA dianas o silenciamiento de la traducción5.
El primer miRNA lin-4, fue descubierto en el nematodo Caenorhabditiselegans en 1993 por Ambros V et al.6. En la actualidad son muchos los trabajos de investigación dirigidos a su identificación y función, por su implicación en diferentes procesos fisiológicos y patológicos como el cáncer.
Cambios en los patrones de expresión de los miRNA están implicados en la aparición de diferentes enfermedades y en su progresión7-10. Asimismo, pueden desarrollar un papel importante en la resistencia a tratamientos con fármacos11.
La relevancia de los miRNA en cáncer, está asociado con incrementos en su expresión y consecuente desregulación de la expresión de los mRNA diana12. Es importante destacar que algunos miRNA están asociados a un tipo de tumor específico, ejemplo de ello es el miRNA-15/16 está asociado a CLL (leucemia linfocítica crónica)13.
En numerosos tipos de cáncer se han observado que los miRNAs desregulados pueden actuar como miRNAs oncogénicos (oncomiR) o miRNA supresores de tumores, basados en su capacidad de reprimir la expresión de genes oncogénicos o supresores de tumores14. El miR-125b puede actuar como oncomiR en la mayoría de canceres hematológicos y como supresor de tumores en tumores sólidos15-16. Esta paradoja se explica por el pequeño tamaño de los miRNA que le permite unirse a diferentes mRNA diana, alguno de los cuales tienen efectos opuestos.
miRNAs extracelulares en cáncer de pulmón, melanoma, cáncer de colon y linfoma folicular
Aunque los miRNAs se expresan en los diferentes tejidos, pueden ser secretados a los fluidos biológicos17 y se pueden cuantificar en plasma libres o asociados a exosomas o en otros fluidos biológicos como saliva y orina18-20.
A nivel práctico, los miRNA en fluidos biológicos son útiles como biomarcadores para detectar pacientes con cáncer, al ser moléculas estables y fáciles de obtener mediante técnicas no invasivas. Los perfiles de expresión de miRNA pueden ser utilizados no solo como pronóstico de cáncer, sino también como selectivo para el tipo de tumor, teniendo en cuenta las variaciones individuales en la expresión de genes y fenotipos tumorales21. Su aislamiento y cuantificación se puede realizar fácilmente por varios métodos de uso rutinario en laboratorios clínicos como es la técnica de PCR.
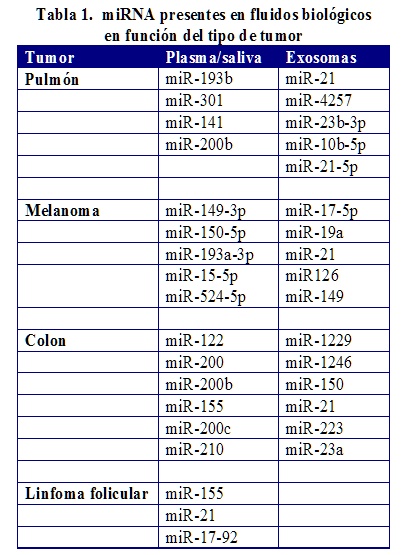
Cambios en los niveles de miRNAs en plasma libre, asociados a exosomas o en saliva se han asociado con diferentes tipos de tumores (Tabla 1), lo cual permite su uso como biomarcadores. Además de diagnóstico puede ser útil para el estudio de la progresión del cáncer.
En cáncer de colon, la presencia en plasma del miRNA-122 y de algunos miembros de la familia miRNA-200 está elevada en pacientes con cáncer no metastásico que presentan una recidiva22. Sin embargo, en estadios de tumores avanzados los miRNA-155 y miRNA-210 son indicativos de resistencia al tratamiento quimioterápico23. Incrementos en la expresión de los miRNAs miR-23a y miR-1246 asociados a exosomas permite la detección de procesos en estadios iniciales y diferenciar sujetos sanos y enfermos19.
Por otro lado, la expresión de los miRNAs let-7g, miR15b, miR-155 y miR-328 en exosomas, pueden ser indicadores de la aparición de recidivas en pacientes con estadios avanzados colon24.
En pacientes con melanoma, se pueden utilizar como biomarcadores los miR149-3p, miR-150-5p, miR193a-5p que no se expresan en pacientes sanos. En cáncer de pulmón, los miR-193b, miR-301, miR-141 y miR-200b están en plasma, mientras que asociados a exosomas se encontraron los miR-23b-3p miR-10b-5p miR-21-5p4.
La saliva al igual que el plasma tiene un gran potencial como fuente de biomarcadores de tumores lejanos de la cavidad oral. Se pueden encontrar proteínas o transcritos de tumores cerebrales, pancreáticos, pulmón, gástricos, esofágicos e incluso de mama, ovario, próstata y leucemia25.
En el cáncer esofágico, al menos se ha determinado la sobreexpresión de cuatro miRNA (miR-144, miR-10b, miR-451, miR-21) frente a controles26. Este mismo hecho ha sido corroborado en el cáncer de páncreas donde (miR-21, miR-23, miR-181a, miR-181b y miR-196a) se encuentran sobre-expresados en pacientes respecto a controles27.
En tumores de la línea mieloide, los microRNAs que han sido identificados regulando la diferenciación de célula B están el miR-155, miR-21 y miR-17-92. El miR-155 es uno de los más estudiados en células B y su desregulación está relacionada con un aumento de la proliferación de las células B, y la regulación de la actividad de factores AKT y está directamente activado por NF-kB22.
El miR21 actúa inhibiendo la expresión de fosfatasas que limitan la actividad de vías de señales como la AKT y MAPK, siendo regulado por el NF-kB24 y ha sido asociado con un mal pronóstico de linfomas28.
La función del miR-17-92 también está asociada con el c-MYC8, 25, 29,, y con otras dianas proapotóticas como el BCL-2. Asimismo, el miR-21 y miR-17-92 aumentan la resistencia a la quimioterapia, aumentan la proliferación e invasión y disminuyen la apoptosis26.
CONCLUSIÓN
Los miRNA obtenidos de fluidos biológicos, suero, plasma, orina o saliva constituyen unos potenciales biomarcadores que pueden ser utilizados para la detección y pronóstico de los enfermos de cáncer.
REFERENCIAS
-
1.- American Cancer Society. Cancer Facts & Figures 2017. Online www.cancer.org/content/dam/cancer-org/research/cancer-facts-and statistics/annual-cancer-facts-and-figures/2017/cancer-facts-and-figures 2017.pdf
2.- Croce CM. Causes and consequences of microRNA dysregulationin cancer.Nat Rev Genet 2009; 10: 704-714.
3.- Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 2004;116(2):281-97.
4.- Liu Q, Yu Z, Yuan S, Xie W, Li C, Hu Z, Xiang Y, Wu N, Wu L, Bai L, Li Y. Circulating exosomal microRNAs as prognostic biomarkers for non-small-cell lung cancer. Oncotarget. 2017 21;8(8):13048-13058.
5.- Mihailescu R. Gene expression regulation: lessons from noncoding RNAs. RNA 2015; 21: 695-696.
6-Ambros V. The functions of animal microRNAs. Nature. 2004 Sep 16;431(7006):350-5.
7.- Ardekani AM, Naeini MM. The Role of MicroRNAs in Human Diseases. Avicenna. J Med Biotechnol. 2010;2(4):161-79.
8- Schwarzenbach H, Nishida N, Calin GA, Pantel K. Clinical relevance of circulating cell-free microRNAs in cancer Nat Rev ClinOncol. 2014;11(3):145-56.
9.- Hayes J, Thygesen H, Tumilson C, Droop A, Boissinot M, Hughes TA, Westhead D, Alder JE, Shaw L, Short SC, Lawler SE. Prediction of clinical outcome in glioblastoma using a biologically relevant nine-microRNA signature. Mol Oncol. 2015;9(3):704-14.
10- Zheng Q, Chen C, Guan H, Kang W, Yu C. Prognostic role of microRNAs in human gastrointestinal cancer: A systematic review and meta-analysis. Oncotarget. 2017. Mar 29.
11- Guo H, Chen Y, Hu X, Qian G, Ge S, Zhang J. The regulation of Toll-likereceptor 2 by miR-143 suppresses the invasion and migration of a subset of human colorectal carcinoma cells. Mol Cancer. 2013; 12:77-87.
12.- Lorio MV, Croce CM. Causes and consequences of microRNA dysregulation. Cancer J. 2012; 18: 215-222.
13- Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K, Rassenti L, Kipps T, Negrini M, Bullrich F, Croce CM. Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sci U S A. 2002;99(24):15524-9.
14.- Oliveto S, Mancino M, Manfrini N, Biffo S. Role of microRNAs in translation regulation and cancer. World J Biol Chem. 2017;8(1):45-56.
15.- Shaham L, Binder V, Gefen N, Borkhardt A, Izraeli S. MiR-125 in normal and malignant hematopoiesis. Leukemia 2012;26:2011-8.
16.- Sun YM, Lin KY, Chen YQ. Diverse functions of miR-125 family in different cell contexts. J Hematol Oncol 2013;6:6.
17.- Kosaka N, Iguchi H, Yoshioka Y et al (2010) Secretory mechanisms and intercellular transfer of microRNAs in living cells. J Biol Chem 285(23):17442-17452.
18.- Lasser C, SeyedAlikhani V, Ekstrom K, Eldh M, Torregrosa Paredes P, Bossios A, Sjostrand M, Gabrielsson S, Lotvall J, Valadi H. Human saliva, plasma and breast milk exosomes contain RNA: uptake by macrophages. J Transl Med 2011;9:9.
19.- Ogata-Kawata H, Izumiya M, Kurioka D, Honma Y, Yamada Y, Furuta K, Gunji T, Ohta H, Okamoto H,Sonoda H, Watanabe M, Nakagama H, Yokota J, Kohno T, Tsuchiya N. Circulating exosomal microRNAs as biomarkers of colon. PLoSOne. 2014 4;9(4)e92921.
20.- Matsuzaki J, Ochiya T. Circulating microRNAs and extracellular vesicles as potential cancer biomarkers: a systematic. 2017 Int J Clin Oncol. 2017;22(3):413-420.
21.- Kozomara A, Griffiths-Jones S. miRBase: integrating microRNA annotation and deep-sequencing data. Nucleic Acids Res. 2011; 39:D152-D157.
22.- Maierthaler M, Benner A, Hoffmeister M, Surowy H, Jansen L, Knebel P, Chang-Claude J, Brenner H, Burwinkel B. Plasma miR-122 and miR-200 family are prognostic markers in colorectal cancer. Int J Cancer. 2017;140(1):176-187.
23.- Chen J, Wang W, Zhang Y, Chen Y, Hu T. Predicting distant metastasis and chemoresistance using plasma miRNAs. Med Oncol. 2014;31(1):799.
24.- Monzo M, Santasusagna S, Moreno I, Martinez F, Hernández R, Muñoz C, Castellano JJ, Moreno J,Navarro A. Exosomal microRNAs isolated from plasma of mesenteric veins linked to liver metastases in resected patients with colon cancer. Oncotarget. 2017; 9;8(19):30859-30869.
25.- Rapado-González Ó, Majem B, Muinelo-Romay L, López-López R, Suarez-Cunqueiro MM. Cancer Salivary Biomarkers for Tumours Distant to the Oral Cavity. Int J Mol Sci. 2016;17(9).
26.- Xie Z, Yin X, Gong B, Nie W, Wu B, Zhang X, Huang J, Zhang P, Zhou Z, Li Z. Salivary microRNAs show potential as a noninvasive biomarker for detecting resectable pancreatic cancer. Cancer Prev Res (Phila). 2015;8(2):165-73.
27.- Humeau M, Vignolle-Vidoni A, Sicard F, Martins F, Bournet B, Buscail L, Torrisani J, Cordelier P. Salivary MicroRNA in Pancreatic Cancer Patients. PLoS One. 2015;10(6):e0130996.
28.- Navarro A, Clot G, Prieto M, Royo C, Vegliante MC, Amador V et al. microRNA expression profiles identify subtypes of mantle cell lymphoma with different clinic biological c haracteristics. Clin Cancer Res 2013; 19: 3121-3129.
29.- Rabinowits G, Gerçel-Taylor C, Day JM, Taylor DD, Kloecker GH. Exosomal microRNA: a diagnostic marker for lung cancer. Clin Lung Cancer. 2009;10(1):42-6.
CORRESPONDENCIA:
Dra. Raquel Alcaraz
Unidad de Investigación,
Hospital Universitario de Burgos.
Burgos. España.
Email: ralcaraz @ hubu.es
Comentario del revisor Daniel Alameda Serrano, PhD. Programa de Tumores Sólidos. Centro de Investigación Médica Aplicada (CIMA), Pamplona. España.
La detección de marcadores genéticos en biopsia líquida está suponiendo una revolución no solo en la detección precoz sino también en la monitorización de la enfermedad en múltiples tipos tumorales. La búsqueda de nuevos biomarcadores que aprovechen esta tecnología es de vital importancia.
Como se comenta acertadamente en la presente revisión, los miRNAs están surgiendo como biomarcadores por su relevancia biológica en cáncer y su estabilidad en biopsia líquida. Su validación en cohortes independientes de pacientes será esencial para validar la utilidad clínica de estas moléculas.
Comentario de la revisora Prof. Victoria Valls i Belles, PhD. Unidad Predepartamental de Medicina. Facultad de Medicina, Universidad de Castellón. España
Los autores realizan una revisión del papel de los miRNA como biomarcador para el diagnóstico y desarrollo del cáncer. La expresión aberrante de estas moléculas constituye una huella genética, algunos de ellos específicos de un tipo determinado de cáncer. Su obtención a partir fluidos biológicos y la metodología para su identifiación y análisis permite ser utilizada como técnica no invasiva para su uso en clínica.
Por ello, en la actualidad son muchos los trabajos de investigación que dirigen sus estudios a identificar estos tipos de miRNAs en fluidos biológicos.
